Small Methods | 一种用于归档和检索医学磁共振成像数据的高效DNA存储系统
近日,中国科学院深圳先进技术研究院姜青山研究员、黄小罗高级工程师、中国农业科学院深圳农业基因组研究所戴俊彪研究员等联合在国际学术期刊Small Methods (IF=12.4)上发表了题为An Effective DNA-Based File Storage System for Practical Archiving and Retrieval of Medical MRI Data的研究文章(图1)。
文章中设计了一种名为"EDS"的DNA存储方法,通过改进编码模型、引入冗余核苷酸和设计索引技术,实现了医学MRI数据的可靠归档和检索。

图1. 文章上线截图
文章链接:https://doi.org/10.1002/smtd.202301585
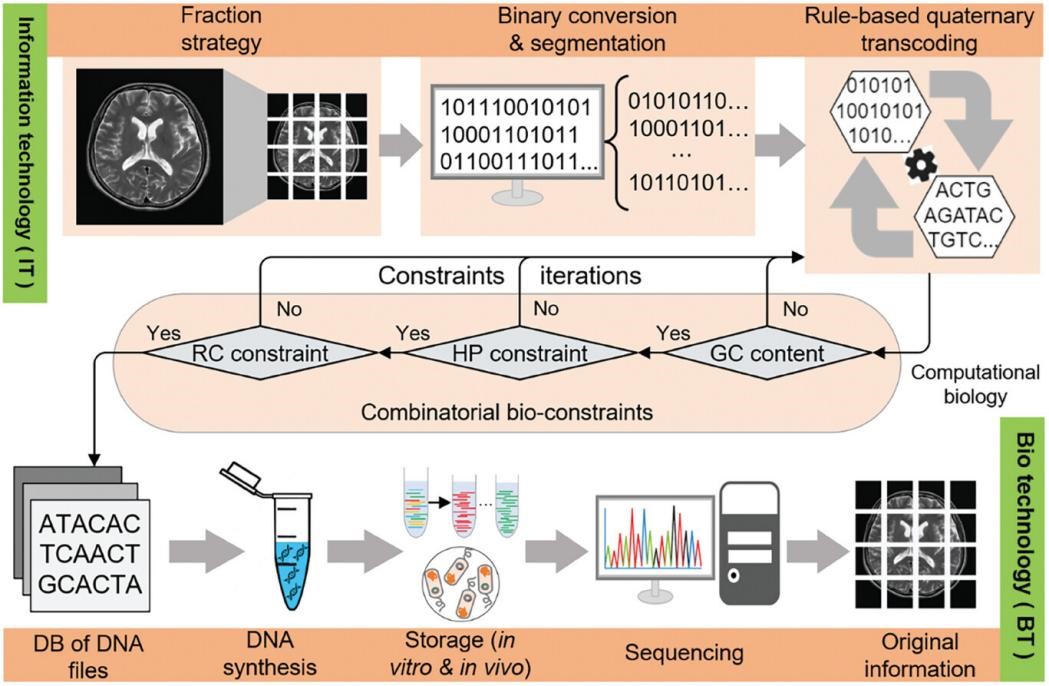
医学MRI数据是诊断、治疗规划和疾病监测等多种领域的重要工具。利用DNA存储技术保存医学MRI数据将有助于人类健康管理。通过DNA存储,可以确保这些重要数据在数千年内安全保存并精确恢复,从而保证了这些重要数据的长期存储。此外,DNA存储还使得过去的健康数据更容易被未来的研究人员获取,这对于纵向研究非常重要,因为它允许研究人员研究疾病的进展和治疗效果。该工作提出了一种名为“EDS”的方法(图2),通过三个关键组成部分实现了医学MRI数据的归档。首先,研究团队设计了一种新颖的分块策略,解决了旋转编码导致的数据丢失问题。其次,提出了一种基于规则的四进制转码方法,满足生化约束条件并确保可靠的数据映射。最后,设计了一种索引技术,简化了随机搜索和访问过程。
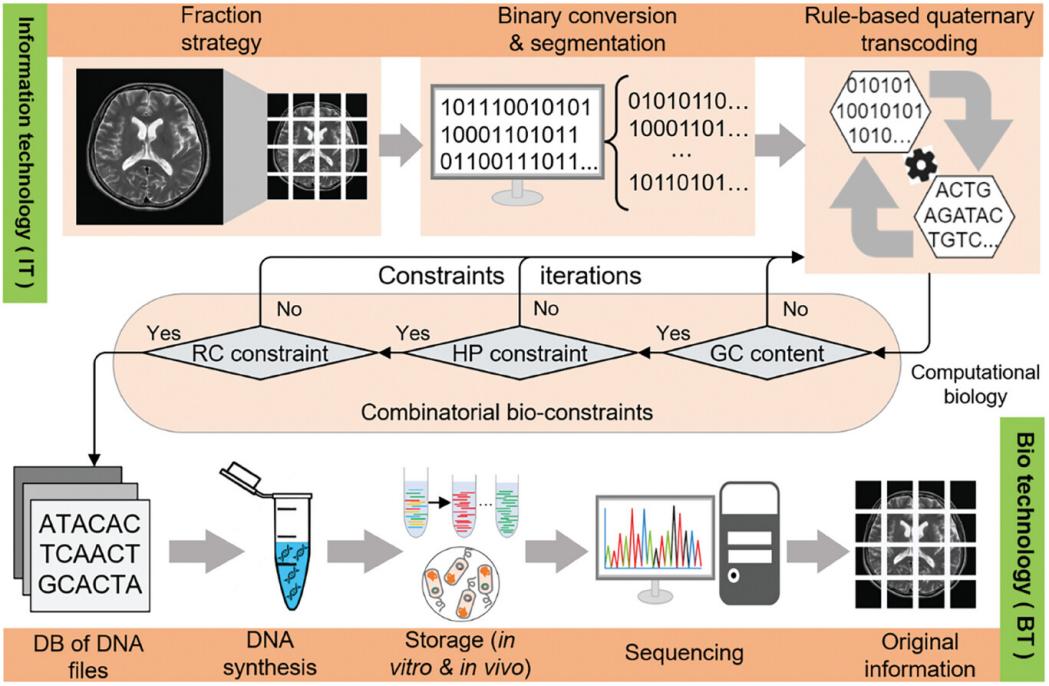
图2. EDS方法流程图
研究团队提出了一种名为“DFS”的索引技术,克服了DNA文件存储中高额外开销的挑战,旨在简化DNA文件存储的组织结构,实现灵活的随机搜索、访问和文件管理。
图3中展示的正则标签(RT,一个碱基对)有效地帮助搜索特定的分块(子图像),将其与其他分块区分开来。图像被分割为16个分块,每个分块被分配了不同的基因标签,序列索引中设计了唯一的地址(4个碱基对),以精确定位和检索所需的序列;此外,在随机有效负载(平均长度为107个碱基对)之前添加了不同的标签(DT,四个碱基对),用于区分每个分块的解码二进制数据,进而简化了存储数据的组织。
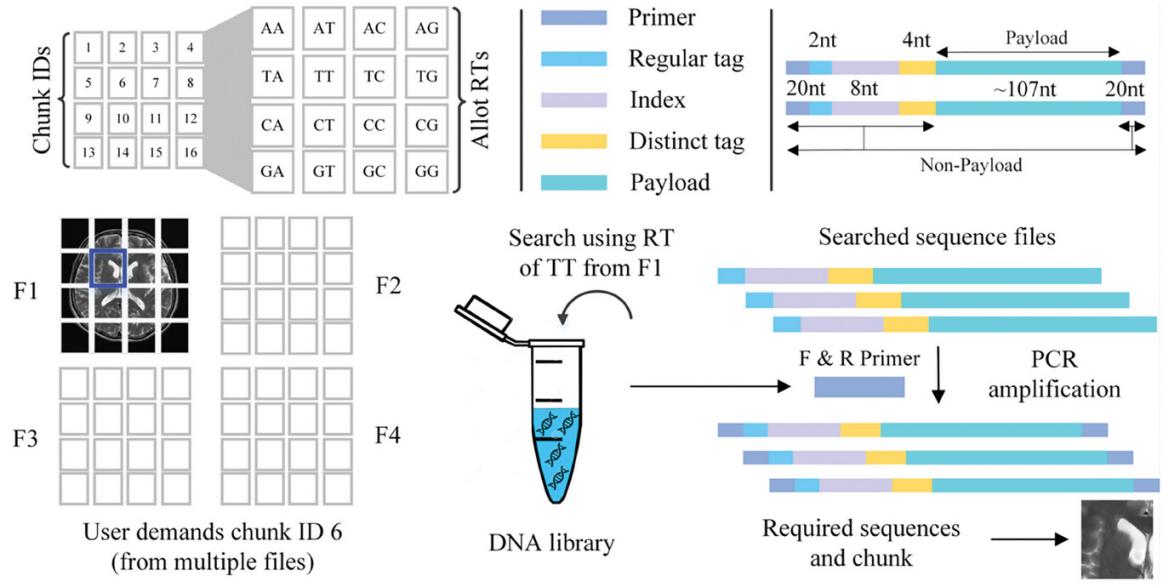
图3. DFS技术示意图
该工作在计算时间上也提出了新的方法,通过利用多进程技术优化DNA存储编码流程,将编码任务分解成多个子任务,并分配多个CPU进行并行计算,提高编码速率,实验共测试了72GB的人体MRI数据,完成编码仅需9个小时,编码时间效率提升明显,同时基于数据进行了预测,1TB量级的数据在120 h能完成。通过计算机模拟和生物合成实验证实,EDS方法在医学MRI数据存储方面表现出色,并且具有更好的生化约束控制和较短的计算时间。为医学MRI数据的DNA存储开辟了新的途径。
图4. 体内合成验证EDS 性能测试
这项研究成果为医学领域的数据存储和检索提供了新的可能性,具有重要的实际应用前景。随着进一步的研究和发展,DNA存储技术有望成为医学数据管理的重要工具,为医学健康领域带来更多的创新和进步。
中国科学院深圳先进技术研究院姜青山研究员、黄小罗研究员高级工程师,中国农业科学院深圳农业基因组研究所戴俊彪研究员为该文章的通讯作者,博士生Abdur Rasool、硕士生洪经纬为论文的共同第一作者。该研究获得国家重点研发项目、深圳市科技项目等多个基金的资助。