Advanced Science | 多模态蛋白表征方法及其迁移性量化
5月30日,中国科学院深圳先进技术研究院数字所生物医学信息中心殷鹏团队在Advanced Science (IF:17.51)在线发表了最新研究成果,题为“A Multimodal Protein Representation Framework for Quantifying Transferability Across Biochemical Downstream Tasks”。该工作提出了一种多模态蛋白质表征方法,通过融合多种蛋白质模态,包括序列、结构和基因本体(GO)信息来实现对蛋白的高效表征。同时,提出了一种基于最优传输的特征空间表示度量,用于量化从预训练的多模态表征到下游任务的动态迁移性。这种度量可以有效地捕捉任务间的分布差异,并预测任务间的适应性。这项研究的成果有助于更好地理解蛋白质的性质和功能,为计算生物学领域的研究提供了新的工具和方法。助理研究员胡帆博士为论文的第一作者,数字所硕士研究生胡奕绅、张维鸿为共同一作。潘毅教授为论文的共同作者,殷鹏副研究员为论文的通讯作者。

文章上线截图
蛋白质是生命的物质基础,是构成细胞的最基本的有机物,担当着生命活动承担者的角色。针对蛋白质的表征学习,简单来说,就是通过计算机算法将蛋白质的复杂信息转化为一种可以被计算机理解和处理的形式,如向量、矩阵等。其意义在于使我们能够利用计算机的强大计算能力来研究和理解蛋白质的复杂性,以及预测蛋白质的行为。大多数现有的蛋白质表示方法都来自于为自然语言文本设计的自监督语言模型。然而,蛋白质的结构和功能是复杂的,且在不同的生物环境中可能会发生变化。因此,如何将蛋白质的序列、结构和功能进行有效融合,以掌握更丰富的多模态表征信息,进而提升下游任务的性能,如蛋白质功能和蛋白-蛋白结合预测等,是一个重要的挑战。另一方面,现有研究表明,下游任务通常可以从预训练模型的信息迁移中受益。那么,是否能量化这种迁移性,从而确定模型的预训练与下游任务间的定量关系以及任务间特征空间的分布与其相互间迁移性的定量关系?解决这些问题对于蛋白表征的训练及应用具有重要意义。
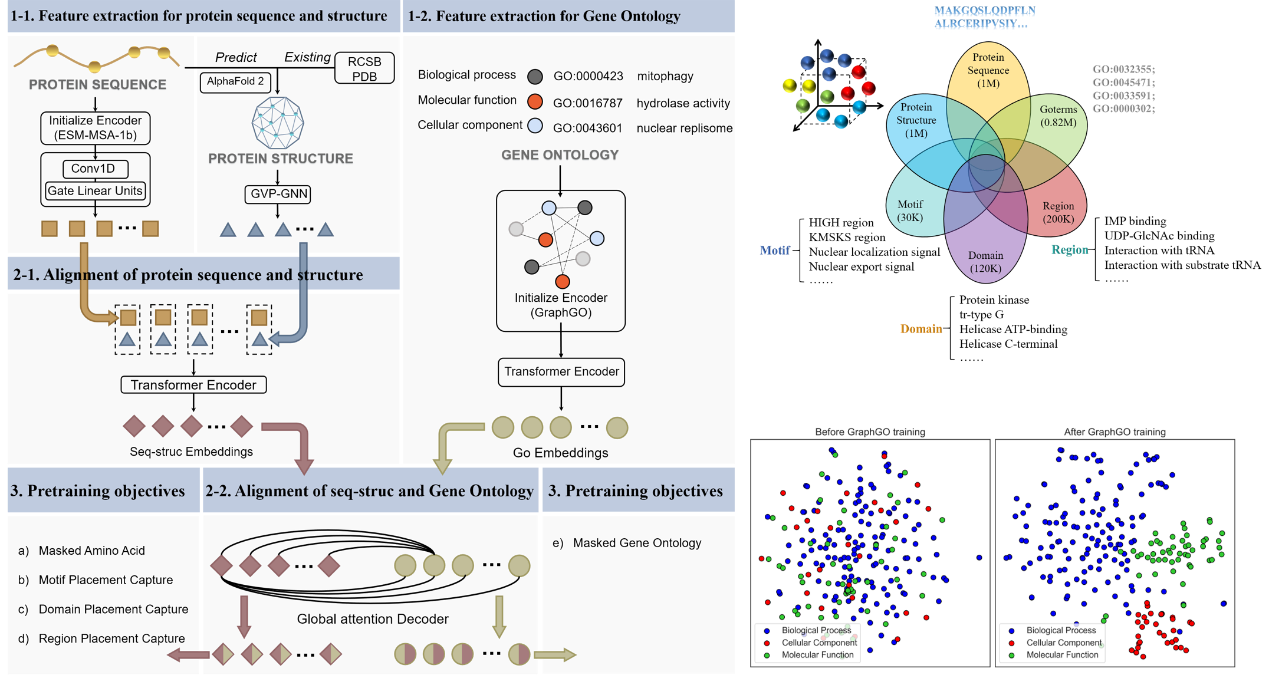
这项工作使用的数据如图1右上所示,包含蛋白序列、结构、功能注释数据以及蛋白细粒度如motif、domain、region等信息。提出的多模态融合表征框架包括四个主要组成部分(图1左):1)蛋白质序列、结构和GO的特征提取。2)通过自注意力机制对蛋白质序列和结构进行token-level的局部对齐。然后将序列-结构特征与GO特征进行全局对齐。3)使用五个特定的预训练目标对多模态模型进行预训练。4)将得到的蛋白质表示应用于下游任务和跨任务学习过程量化。
该方法得到的蛋白多模态表征在多项蛋白相关的下游任务中取得了优异表现,如蛋白稳定性预测、蛋白-蛋白互作预测等。另一方面,这项工作提出了一种新的跨任务迁移性度量方法(OTFRM),用于量化从预训练表征到相关下游任务以及下游任务间相互的动态迁移性。研究者计算了这些下游任务之间的成对距离,并观察到了任务间特征空间分布和适应性之间的强相关性(图2)。该度量方法可用于评估跨任务学习过程,预测适应性,引导各种任务的微调,并指导蛋白质表征学习的神经网络和训练目标设计。
该研究的成果可应用于多类蛋白相关的下游任务包括蛋白性质和功能预测、蛋白-蛋白互作预测、蛋白-药物互作预测等。并且,提出的迁移性度量方法有助于提高预训练模型在特定下游任务的性能,具有广泛的应用前景。
该研究得到了中国科学院战略优先研究计划、国家自然科学基金委、广东省科技厅、深圳市科创委等科技项目的资助。
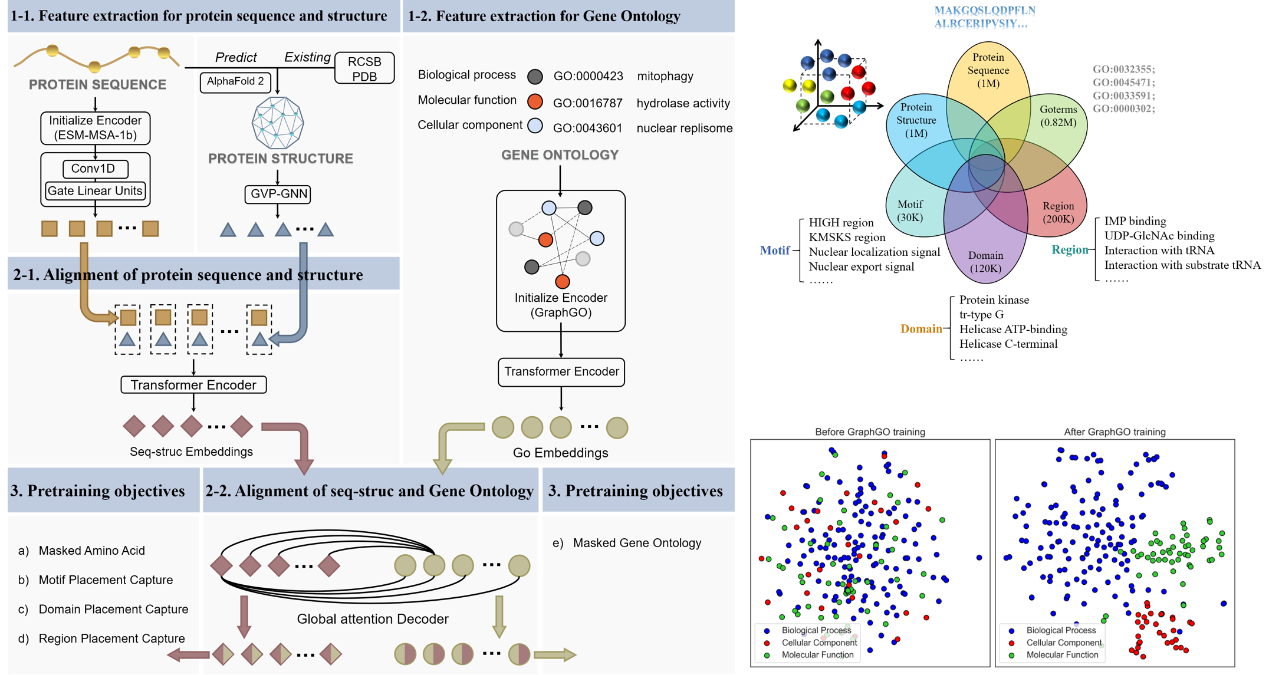
图1. 多模态蛋白表征框架及数据

图2. 跨任务迁移性度量方法