Nucleic Acids Research | 底盘噬菌体的高通量制备
北京时间12月14日,中国科学院深圳先进技术研究院马迎飞团队在国际学术期刊《核酸研究》(Nucleic Acids Research,IF=19))上发表了题为Genome-scale top-down strategy to generate viable genome-reduced phages的文章。该工作报道了一种高通量制备底盘噬菌体的方法,在鉴定噬菌体非必需基因的同时,获得底盘噬菌体,并得到比野生型噬菌体侵染能力更强的基因组简化噬菌体,在噬菌体治疗和噬菌体合成生物学中具有巨大的潜在价值。
深圳先进技术研究院马迎飞研究员为该文章的通讯作者,其博士生袁盛建为文章第一作者。

文章上线截图
噬菌体是一种病毒,能够侵染并杀死细菌。因此,噬菌体在被发现伊始就被用来对抗细菌感染。2020年,马迎飞团队成功联合深圳人民医院开展了深圳市第一例应用噬菌体治疗耐药鲍曼不动杆菌肺部感染的临床试验。噬菌体是地球上多样性最高和最丰富的生物体,也是合成生物学研究中重要的模式生物。底盘噬菌体通过删除基因组上的冗余基因,可以扩大噬菌体颗粒的DNA包装空间,搭载更多的功能基因,增强其在各种生物工程应用中的抗菌活性和潜力,同时增加对噬菌体的理解。
目前,如何高通量删除噬菌体冗余基因对科研人员来说仍然具有挑战性。第一个挑战是怎样能够高通量识别噬菌体的非必需基因?因四分之三的噬菌体基因组无法找到其同源序列,使其不能通过信息分析进行非必需基因判断。第二个挑战是怎样筛选具有生长优势的突变噬菌体?有些噬菌体基因删除并不会使噬菌体“死亡”,但删除会严重影响噬菌体的扩增(准必需基因)。另外第三个挑战是如何在逐删除基因组不同区域这种费力且耗时的情况下,同时筛选能够维持稳健生长的突变噬菌体?
CiPGr方法的开发
CRISPR是细菌对抗噬菌体的防御系统,当噬菌体侵染的细菌含有人工设计的CRISPR系统时,就有一定概率可以删除噬菌体的某个基因(突变株1)。如果用突变株1侵染含有另一个人工设计的CRISPR系统宿主菌时,就有一定概率可以删除噬菌体的另一个基因(突变株2),这样持续下去,就有能得到一个底盘噬菌体(图1)。科研团队猜想,是否可以把所有的含有不同人工设计的CRISPR系统宿主菌混合在一起,让噬菌体基因高通量删除并通过优胜劣汰筛选出侵染能力强的突变株呢?
基于这个启发,团队开发了一种自上而下的全基因组简化方法,称为基于CRISPR-Cas9的迭代噬菌体基因组简化方法(CRISPR/Cas9-based iterative phage genome reduction, CiPGr)。针对所有目标基因,设计不同的CRISPR系统,并在DNA芯片上合成,制备出删除噬菌体不同基因的CRISPR细菌文库。在CiPGr过程中,用一群噬菌体侵染细菌文库,使不同的噬菌体删除不同的基因,形成一个噬菌体突变文库。如果被删除的基因对噬菌体扩增不是必需的,则突变噬菌体后代可以继续侵染新的细菌。噬菌体突变文库不断转接到新鲜培养的细菌文库中,使得基因缺失在噬菌体基因组中不断积累,而具有生长优势的基因组简化噬菌体将在种群中占主导地位,最终被富集并更可能被分离纯化出来(图2)。
非必需基因的鉴定
团队使用CiPGr成功简化了四种不同的有尾噬菌体(T7、T4、seszw和selz)基因组,得到了它们的底盘噬菌体、非必需基因集以及比野生型侵染能力更强的基因组简化噬菌体。
对获得的噬菌体突变文库进行二代基因组测序,团队通过生物信息分析,即可操作简单、高效率的获得到噬菌体的可删除基因集。可删除基因集中的基因数量在4个不同噬菌体中,占总基因数的50%左右。团队根据实验结果将噬菌体基因分为三类:(1)非必需基因:噬菌体突变文库中的删除丰度>5%的基因,这些基因在分离的60个单克隆突变噬菌体基因组中没有被删除;(2)准必需基因:噬菌体突变文库中删除丰度<5%基因,这些基因的删除严重影响了噬菌体的扩增,导致在连续转接过程中,种群中相应突变体的快速淘汰;(3)必需基因:在可删除基因集中未检测到的基因(图3)。
最佳裂解能力简化噬菌体鉴定
在对突变噬菌体的表型进行鉴定时,团队发现部分突变株的抑菌或杀菌能力比野生型强,因此,科研人员设计了实验对这些更强杀菌能力的突变株进行分离与鉴定,并将获得的噬菌体突变文库与野生型宿主菌混合在一起进行培养。经过8次连续转接后,具有较高侵染效率的突变株能产生更多子代,使其在种群中占主导地位,从而分离出强杀菌能力的简化基因组噬菌体。其中T4、seszw和selz的杀死同样数量细菌的时间比野生型减少了一半以上(图4),由此说明通过基因组简化可以获得比野生型噬菌体裂解能力更强突变株的可行性。
最简化基因组噬菌体筛选
在对CiPGr生成的单克隆突变噬菌体进行测序时,科研团队观察到基因缺失数量随着转接次数的增加而在单克隆突变噬菌体基因组中不断积累。CiPGr应用于四种不同的有尾噬菌体(模式噬菌体T7和T4;沙门氏菌噬菌体seszw和selz),导致这些噬菌体的突变体删除8-23%(3.3-35 kbp)的序列,产生了最简底盘噬菌体。持续的转接过程使具有较大生长优势的突变株将在突变库中占主导地位,并在众多突变株中有更大概率进入下一轮转接过程。因此,单克隆突变噬菌体基因组中基因删除的顺序可能表明这些基因在噬菌体扩增中的重要性顺序,即基因越早被删除,该基因对噬菌体扩增就越不重要(图5)。
在CiPGr的操作过程中,研究团队只需要获得噬菌体基因组序列,就能够将该方法轻松推广至其他野生型噬菌体而不需要其他先验的知识。同时,这些简化噬菌体能够整合更多的基因元件以增强噬菌体的功能,在噬菌体治疗中具有巨大的应用潜力。此外,噬菌体基因组中必需、非必要和准必需基因的鉴定对于噬菌体基因组的重新设计和噬菌体生物学的研究具有重大意义。
该研究得到了科技部重点研发计划、中科院先导B、中科院定量工程生物学重点实验室、广东省合成基因组学重点实验室及深圳合成生物学创新研究院的资助。
参考文献
1. Yuan S, Shi J, Jiang J, Ma Y. Genome-scale top-down reduction of phages to generate viable minimal phage genomes. Nucleic Acids Research; 2022, DOI: 10.1093/nar/gkac1168.
2. Tan X, Chen H, Zhang M, Zhao Y, Jiang Y, Liu X, Huang W, Ma Y. Clinical Experience of Personalized Phage Therapy Against Carbapenem-Resistant Acinetobacter baumannii Lung Infection in a Patient With Chronic Obstructive Pulmonary Disease. Front Cell Infect Microbiol. 2021 Feb 26;11:631585. doi: 10.3389/fcimb.2021.631585. PMID: 33718279; PMCID: PMC7952606.
3.Peifen Chen, Ziqiang Liu, Xin Tan, Haijiang Wang, Yiyang Liang, Yingjun Kong, Wenxue Sun, Liqin Sun, Yingfei Ma, Hongzhou Lu. Bacteriophage therapy for empyema caused by carbapenem-resistant Pseudomonas aeruginosa. BioScience Trends; 2022, Volllume16, Issue 2, doi.org/10.5582/bst.2022.01147.

图1 噬菌体基因连续删除示意图
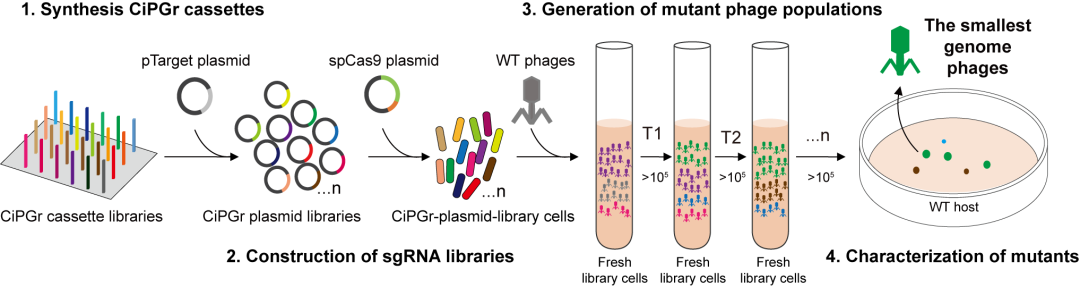
图2 实验流程设计
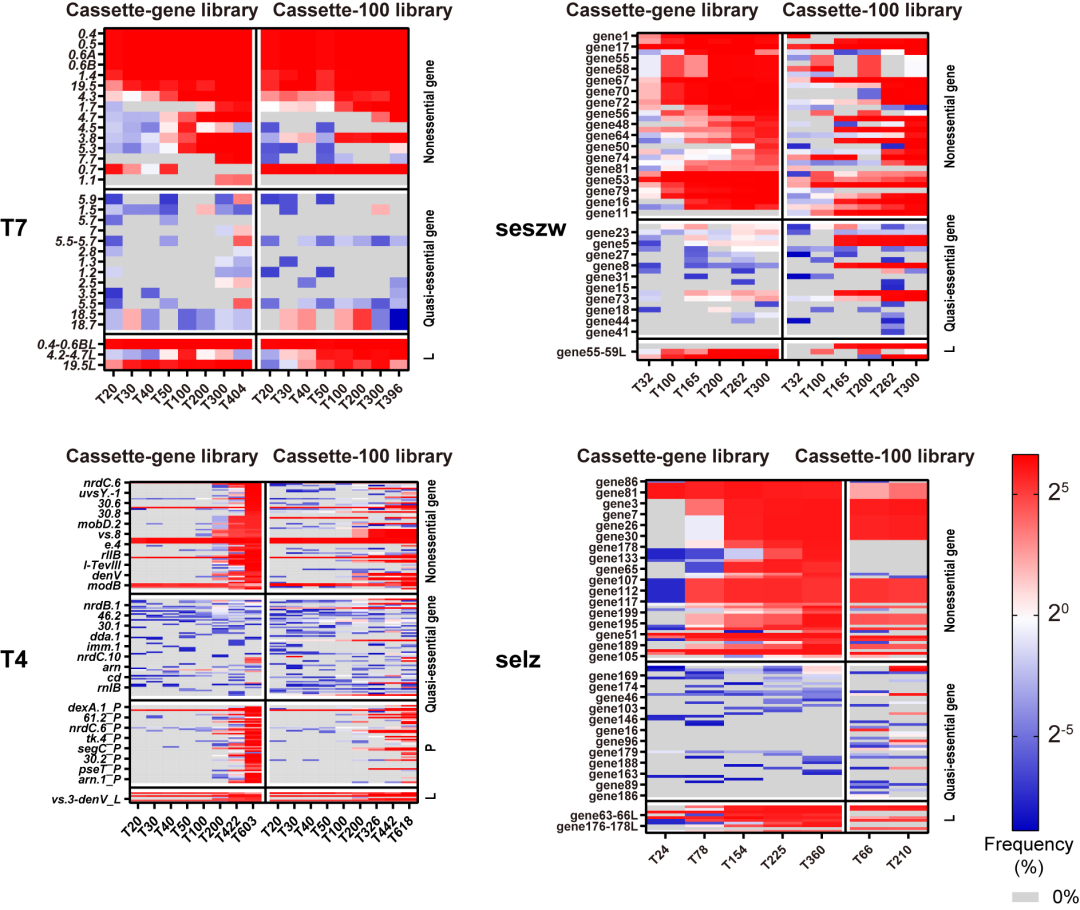
图3 噬菌体的基因删除与分类

图4 比野生型噬菌体更强突变株的筛选(A)和鉴定(B)
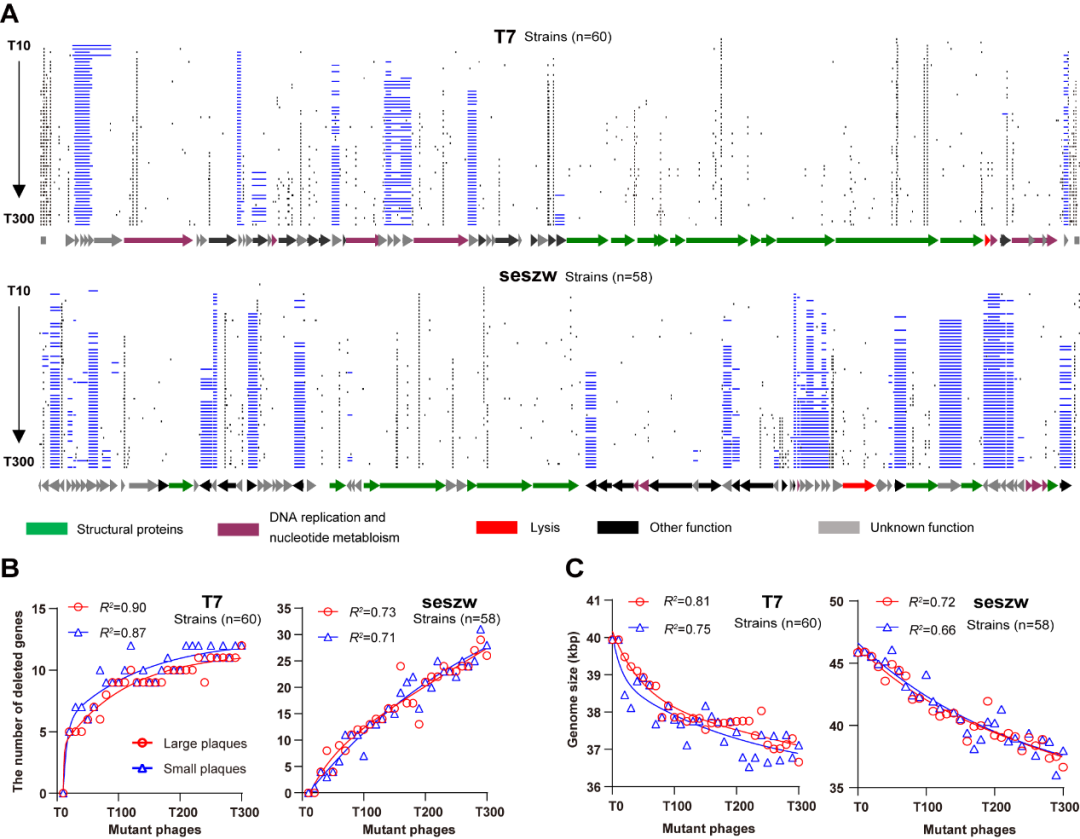
图5 单克隆突变噬菌体的基因删除




