Mol. Syst. Biol. | 刘陈立/傅雄飞开发空间连续定向进化系统
9月21日,中国科学院深圳先进技术研究院刘陈立课题组与傅雄飞课题组合作在Molecular Systems Biology上发表了题为“Exploiting spatial dimensions to enable parallelized continuous directed evolution”的文章。该项工作定量研究了细菌-噬菌体在空间上共同生长迁移的动力学过程,并基于这一定量理解,发展了空间噬菌体辅助连续定向进化系统(Spatial Phage-Assisted Continuous Evolution, SPACE),实现了合成生物元件的大规模平行进化。
深圳先进院合成所刘陈立研究员、傅雄飞研究员为该文章的通讯作者,助理研究员魏婷、赖旺生、博士研究生陈茜、及助理研究员张易为该文章的共同第一作者。

文章上线截图
文章链接:https://doi.org/10.15252/msb.202210934
合成生物学旨在以工程化的理念改造或创造人工生物系统,以期理解生命、改造生命、合成生命乃至设计生命。生物元件作为合成生物系统的基本单元,它的数量和功能制约着合成生物学的发展。传统手段通过挖掘天然生物系统获得的生物元件,其性能常常不能满足人们的需求;通过理性设计发展全新生物元件的方式虽有潜力,但目前还受限于设计能力;而通过定向进化,利用进化这个“上帝之手”的力量来对已有元件进行改造和优化获得所需的功能,已成为合成生物学领域中一项重要的关键平台性技术。
传统的定向进化方法一般分为突变建库和筛选这两个步骤[1],循环迭代需要大量的重复操作,耗费人力物力和时间。连续定向进化方法(continuous directed evolution)则利用可自我复制的生物体,在其基因组复制过程引入突变、并利用突变后该生物体复制扩增能力的差异性变化来实现建库与筛选这两个步骤的自动连接和迭代循环,从而减少人力劳动,使定向进化快速进行[2]。2011年,美国哈佛大学David Liu实验室开发了噬菌体辅助连续进化(Phage-Assisted Continuous Evolution, PACE)[3],将M13噬菌体的增殖与待进化的生物分子功能关联在一起,在一天内就实现了数十轮的定向进化,大大提升了进化效率,是定向进化技术发展中里程碑式的重要突破。PACE已被应用于RNA聚合酶、TALEN、Cas9、碱基编辑器等重要酶类的进化[3-6]。
然而,如果我们要同时进化多个目标蛋白,目前还缺少一种简便的定向进化技术/方法;另一方面,PACE系统需要连续培养装置(恒化器,chemostat)、复杂的流速控制与检测设备和一定的操作技巧,因此普通实验室不太容易开展PACE实验。新发展的SPACE系统将复杂的进化装置简化成一个每个生物实验室都有的琼脂平板,很好的解决了这两个难题。
定量解析宿主-病毒共迁移系统的进化时空动力学
SPACE系统的开发灵感来源于刘陈立课题组前期关于细菌迁移定植的研究(详见:专家点评Nature | 刘陈立/华泰立合作利用迁徙进化实验揭示合成生物建构原理)[7]。在前期研究中,团队发现处在空间扩张前锋(front)的细菌始终保持在对数生长期,而落后于前锋的细菌不再进行定向迁移运动并继续生长进入平台期。这不就是一个“移动的恒化器”吗(图1A)?
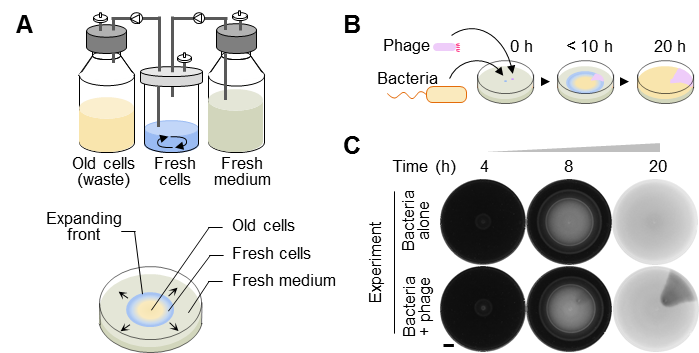
图1 细菌空间扩展过程中的噬菌体感染
A,细菌空间扩展系统与连续培养装置的相似性;B,细菌-噬菌体共迁移实验示意图;C,实验结果
本项目研究团队原创性地利用这个“移动的恒化器”替代PACE系统中噬菌体的持续扩增所需要的连续培养装置,从而大幅简化了系统。研究团队构建了细菌-噬菌体共迁移实验体系,使得原本没有运动能力的噬菌体被处于空间扩张运动过程中的宿主细菌携带并广泛传播。由于被感染的细菌生长速率慢于未被感染的细菌,最终在软琼脂表面的菌苔上产生肉眼可见的低细菌密度的扇形感染区(图1BC)。虽然宿主-病毒共迁移系统在自然界普遍存在,但人们对迁移过程中病毒如何进化了解的很少。
为了理解这一过程,刘陈立课题组与傅雄飞课题组合作,以经典的传染病模型SIR(易感者Susceptible,感染者Infected,康复者Recovered)为基础,结合细菌空间扩张运动模型,建立了空间扩张系统的宿主感染模型(Range Expansion with Susceptible Infected Recovered kinetics)(图2)。该数学模型刻画了空间扩张体系下细菌-噬菌体共迁移的时空动力学,解释了扇形噬菌体感染区的形成过程(图1C)。结合模型模拟与定量实验,研究团队发现扇形感染区的面积与噬菌体感染并产生子代的能力呈正相关关系,这使噬菌体感染能力的强弱有了直观的可视化表征方式。
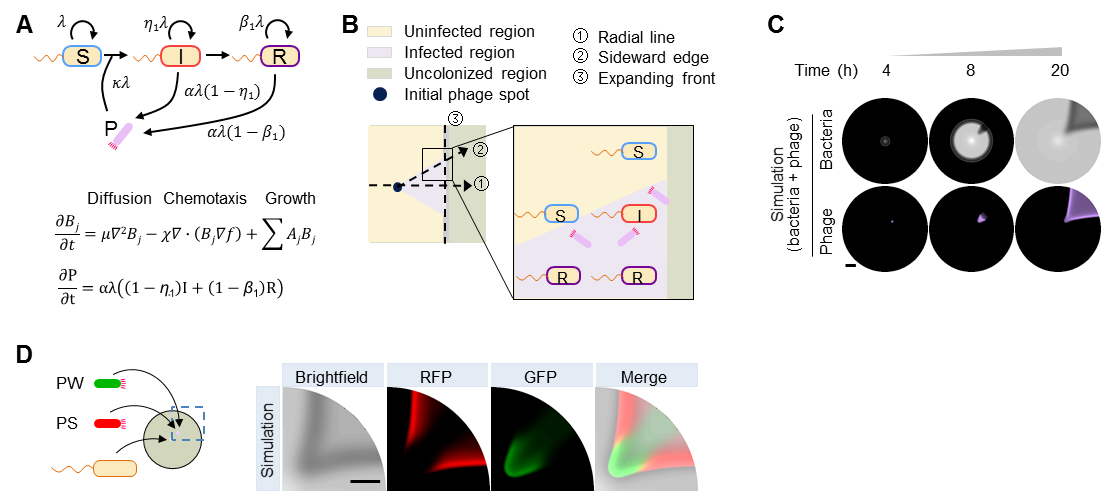
图2 空间扩展过程中噬菌体与细菌互作动力学模型
A,细菌SIR三种状态的转化及噬菌体生产的数学描述;B,感染动态在不同位置和方向上的差异性;C,模型模拟噬菌体感染区的形成动态;D,模型模拟强弱噬菌体初始比例为PS:PW=1:105 的空间竞争
随后,研究团队利用理论模型结合实验进化的方式,解析细菌迁移过程中噬菌体感染宿主的进化动力学。对于宿主而言,它们的进化主要发生在扩展运动的前锋中,这种现象被称为空间分选进化(spatial sorting)[8]。研究团队一开始认为病毒的进化也主要发生在同样的位置。然而,后续研究中团队观察到了与该预期截然不同的结果:与更高的感染能力相关的噬菌体有益突变快速积累的位置并不是在宿主运动的前锋中,而是沿着扇形感染区域的侧边沿。该现象产生的原因与侧边沿处存在的非平衡的感染状态有关。这一发现可能也适用于其它生态系统的宿主/病毒共迁徙现象。
理性设计构建空间连续定向进化系统
基于上述定量理解,团队设计构建了SPACE系统(图3)。为了适应SPACE在半固体培养基中的应用,对宿主菌携带的一个关键诱变质粒进行了功能改造,使具有诱变效果的基因只在宿主细菌被噬菌体感染之后表达。未进化的初始噬菌体感染能力很弱,几乎不能在宿主细菌的空间扩展过程中形成可见的扇形感染区。如果诱变过程中其携带的待进化基因产生了有益突变,则可以产生感染能力增强的子代噬菌体,从而使其感染区域面积显著扩大。
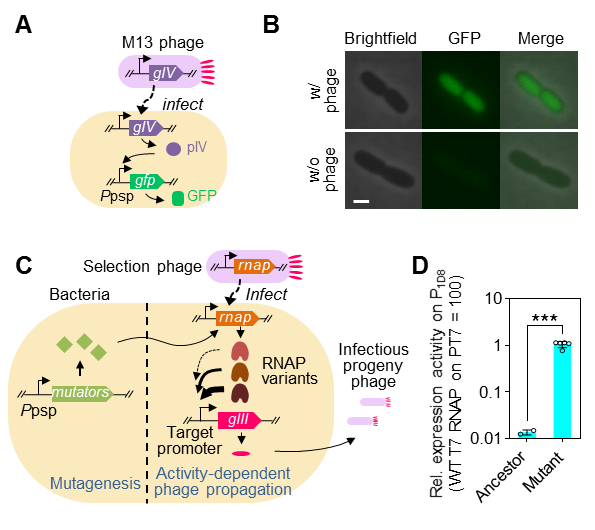
图3 SPACE系统的构建
A, 利用噬菌体休克蛋白(phage shock protein, psp)启动子改造诱变模块;B, psp启动子仅在存在噬菌体感染的条件下启动表达;C,SPACE系统基因线路设计;D,T7 RNA聚合酶进化前后识别目标启动子的活性变化
SPACE实现大规模平行定向进化
SPACE系统具有可视化、简便的优势,可以实现大规模平行进化实验。研究团队利用该系统平行进化了T7 RNA聚合酶识别随机启动子序列的能力(图4),获得了一系列启动子变体-RNA聚合酶突变库,可用于进一步改造及工程应用。
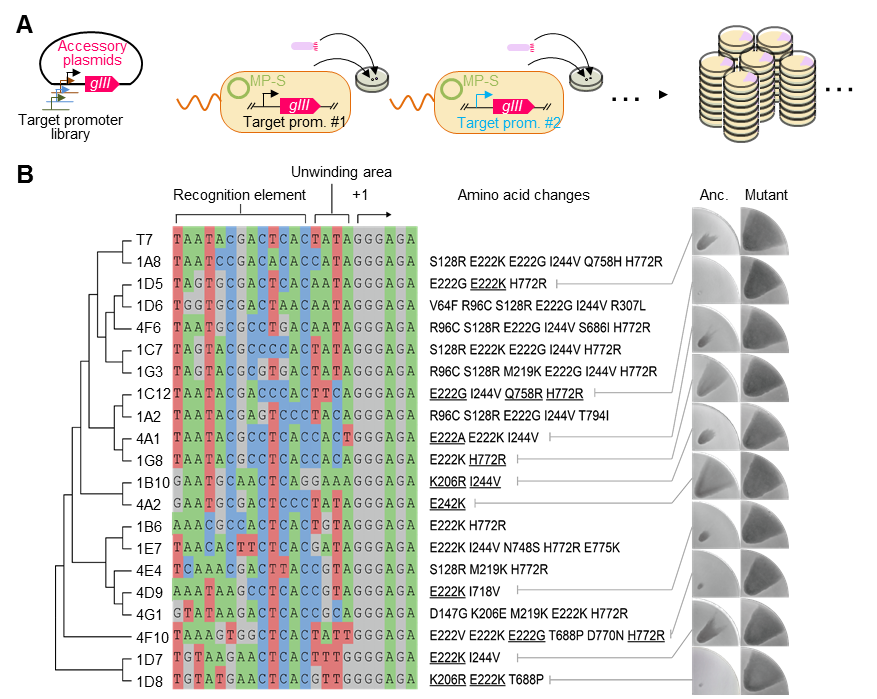
图4 利用SPACE进行大量平行进化
A,平行进化T7 RNA聚合酶对人工启动子库识别活性的实验示意图;B,进化获得的能够识别目标启动子的聚合酶突变体
本项工作着眼于合成生物学研究中可用元件匮乏这一亟待解决的关键问题,在现有连续定向进化方法的基础上,通过实验与模型模拟相结合的方式定量研究了宿主细菌的空间迁移运动对其噬菌体进化的作用规律,并由此利用空间维度发展了蛋白质等生物分子的连续定向进化方法,示范了定量合成生物学的研究范式。SPACE系统可以在普通实验室中实现生物元件的大规模平行进化改造,为合成生物学在化工、医疗等领域的应用提供丰富的元件库。同时,细菌空间迁移运动对噬菌体进化的作用规律,也将对于研究更高等的生物如昆虫、鸟类的迁徙对其携带的可能对人类健康也存在影响的病毒如何进化等生态学理论或调查研究提供参考和示范。
该项工作获得了中科院先导专项B、科技部重点研发计划、国家自然科学基金、中科院交叉学科创新团队等项目支持。
[1] Bloom JD, Arnold FH. In the light of directed evolution: Pathways of adaptive protein evolution [J]. Proceedings of the National Academy of Sciences, 2009, 106(supplement_1): 9995-10000.
[2] Badran AH, Liu DR. In vivo continuous directed evolution [J]. Curr Opin Chem Biol, 2015, 24(1-10.
[3] Esvelt KM, Carlson JC, Liu DR. A system for the continuous directed evolution of biomolecules [J]. Nature, 2011, 472(7344): 499-503.
[4] Hubbard BP, Badran AH, Zuris JA, Guilinger JP, Davis KM, Chen L, Tsai SQ, Sander JD, Joung JK, Liu DR. Continuous directed evolution of DNA-binding proteins to improve TALEN specificity [J]. Nat Methods, 2015, 12(10): 939-942.
[5] Miller SM, Wang T, Randolph PB, Arbab M, Shen MW, Huang TP, Matuszek Z, Newby GA, Rees HA, Liu DR. Continuous evolution of SpCas9 variants compatible with non-G PAMs [J]. Nat Biotechnol, 2020, 38(4): 471-481.
[6] Thuronyi BW, Koblan LW, Levy JM, Yeh WH, Zheng C, Newby GA, Wilson C, Bhaumik M, Shubina-Oleinik O, Holt JR, Liu DR. Continuous evolution of base editors with expanded target compatibility and improved activity [J]. Nat Biotechnol, 2019, 37(9): 1070-1079.
[7] Liu W, Cremer J, Li D, Hwa T, Liu C. An evolutionarily stable strategy to colonize spatially extended habitats [J]. Nature, 2019, 575(7784): 664-668.
[8] Shine R, Brown GP, Phillips BL. An evolutionary process that assembles phenotypes through space rather than through time [J]. Proceedings of the National Academy of Sciences, 2011, 108(14): 5708-5711.




